모델이 너무 간단하면 과소적합(높은 편향) 너무 복잡하면 훈련 데이터에 과대적합(높은 분산)이 될 수 있다.
적절한 편향-분산 트레이드오프를 찾기 위해서는 모델을 주의깊게 평가하여야 한다.
하이퍼 파라미터를 튜닝하고 비교하는 과정을 모델 선택이라고 한다.
-> 주어진 분류 모델에서 튜닝할 파라미터(또는 하이퍼 파라미터)의 최적값을 선택해야 하는 것을 의미
모델 선택에서 같은 테스트 데이터셋을 반복해서 재사용하면, 훈련 데이터셋의 일부가 되는 셈으로 모델이 과대적합된다.
즉 모델 선택을 위한 테스트 데이터셋 사용은 좋은 머신 러닝 방식이 아니다.
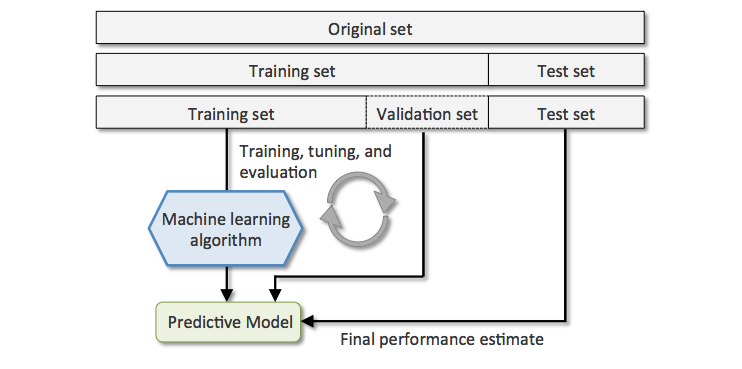
홀드 아웃 교차 검증(Holdout cross-validation)
데이터셋을 훈련 데이터셋, 검증 데이터셋, 테스트 데이터셋으로 나누는 것이 좋다.
(검증 데이터셋을 모델 선택하는데 사용) -> 테스트 데이터셋을 모델이 만나지 못했기 때문에 새로운 데이터에 대한
일반화 능력을 덜 편향되게 추정할 수 있다.
검증 데이터셋을 사용하여 반복적으로 다른 파라미터 값에서 모델을 훈련한 후, 성능을 평가한다.
만족할 만한 하이퍼파라미터 값을 얻었을 경우, 테스트 데이터셋에서 모델의 일반화 성능을 추정한다.
홀드 아웃 방식은 훈련 데이터를 훈련 데이터셋과 검증 데이터셋으로 나누는 방법에 따라 성능 추정이 민감할 수 있다.
검증 데이터셋의 성능 추정이 어떤 샘플을 선택하느냐에 따라서 달라진다.
k-겹 교차 검증은 이를 보안한 좀 더 안정적인 성능 추정 기법이다.
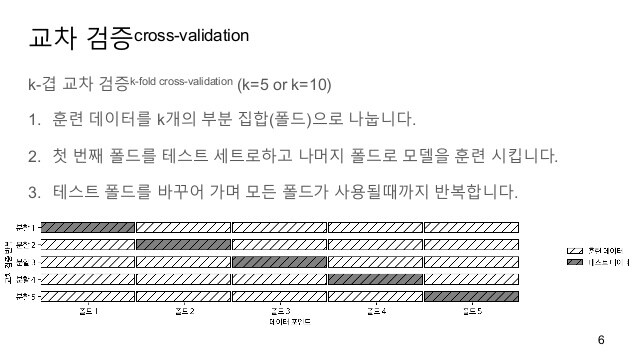
k-겹 교차 검증(k-fold cross-validation)
k-겹 교차 검증에서는 중복을 허용하지 않고 훈련 데이터셋을 k개의 폴드(fold)로 랜덤하게 나눈다.
k-1개의 폴드로 모델을 훈련하고, 나머지 하나의 폴드로 성능을 평가한다.
이 과정을 k번 반복하여 k개의 모델과 성능 추정을 얻는다.
그 다음 서로 다른 독립적인 폴드에서 얻은 성능 추정을 기반으로 모델의 평균 성능을 계산한다.
홀드 아웃 방법에 비해 훈련 데이터셋의 분할에 덜 민감한 성능 추정을 얻을 수 있다.
일반적으로 k-겹 교차 검증을 사용하며 테스트 데이터셋에서 모델의 성능을 평가할 때,
만족할 만한 일반화 성능을 내는 최적의 하이퍼파라미터 값을 찾기 위해 사용한다.
만족스러운 하이퍼 파라미터 값을 찾은 후에는 전체 훈련 데이터셋을 사용하여 모델을 다시 훈련한다.
이후, 독립적인 테스트 데이터셋을 사용하여 최종 성능을 추정한다.
k-겹 교차 검증이 중복을 허용하지 않는 리샘플링 기법이기 때문에 모든 샘플 포인트가 훈련하는 동안
테스트 폴드로 검증에 딱 한 번 사용되는 장점이 있다.
따라서 홀드아웃 방법보다 모델 성능의 추정에 분산이 낮다.
또한 각 폴드의 추정 성능 E(분류 정확도 또는 오차 등)을 이용하여 모델의 평균 성능을 계산한다.
k값이 증가하면 더 많은 훈련 데이터가 각 반복에 사용되고 모델 성능을 평균하여 일반화 성능을 추정할 때,
더 낮은 편향을 만든다. k 값이 아주 크면 교차 검증 알고리즘의 실행 시간이 늘어나고 분산이 높은 추정을 만든다.
(훈련 폴드가 서로 많이 유사해지기 때문에) k가 작으면 모델을 학습하고 평가하는 계산 비용이 줄어든다.
-> 비교적 작은 훈련 데이터셋에는 k를 늘이는 것이 좋다. k의 기본값으로 10이 적절하며, 대규모 데이터셋으로 작업할 떄는
k=5를 사용하는 것이 좋다.
LOOCV(Leave-One-Out Cross-Validation)LOOCV는 k-겹 교차 검증 방법중에 하나로 폴드의 개수가 훈련샘플의 개수가 같다.
(하나의 샘플이 각 반복에서 테스트로 사용된다.)
아주 작은 데이터셋을 사용할 때 권장된다.
from sklearn.model_selection import LeaveOneOut
계층적 k-겹 교차 검증(stratified k-fold cross-validation)
k-겹 교차 검증보다 더 나은 편향과 분산 추정을 만드는 방법이다.
특히 클래스 비율이 동등하지 않을 때 유용하다.
계층적 k-겹 교차 검증은 각 폴드에서 클래스 비율이 전체 훈련 데이터셋에 있는 클래스 비율을 대표하도록 유지한다.
StratifiedKFold클래스의 shuffle 매개변수를 True로 지정하면 폴드를 나누기 전에 샘플을 섞는다.
또한 이 때, random_state를 지정하면, 에러가 발생한다.(클래스 비율을 유지해야 하기 때문에)
shuffle의 default 값은 False이다.
import numpy as np
from sklearn.model_selection import StratifiedKFold
kfold=StratifiedKFold(n_splits=10).split(X_train, y_train)
scores=[]
for k, (train, test) in enumerate(kfold):
pipe_lr.fit(X_train[train], y_train[train])
score=pipe_lr.score(X_train[test], y_train[test])
scores.append(score)
print('폴드: %2d, 클래스 분포: %s, 정확도: %.3f'%(k+1, np.bincount(y_train[train]), score))
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 1, 클래스 분포: [256 153], 정확도: 0.935
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 2, 클래스 분포: [256 153], 정확도: 0.935
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 3, 클래스 분포: [256 153], 정확도: 0.957
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 4, 클래스 분포: [256 153], 정확도: 0.957
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 5, 클래스 분포: [256 153], 정확도: 0.935
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 6, 클래스 분포: [257 153], 정확도: 0.956
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 7, 클래스 분포: [257 153], 정확도: 0.978
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 8, 클래스 분포: [257 153], 정확도: 0.933
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 9, 클래스 분포: [257 153], 정확도: 0.956
Pipeline(steps=[('standardscaler', StandardScaler()),
('pca', PCA(n_components=2)),
('logisticregression', LogisticRegression(random_state=1))])
폴드: 10, 클래스 분포: [257 153], 정확도: 0.956
print('\nCV 정확도: %.3f +/- %.3f' %(np.mean(scores), np.std(scores)))
cross_val_score를 이용한 k-겹 계층별 교차 검증 함수
from sklearn.model_selection import cross_val_score
scores=cross_val_score(estimator=pipe_lr, X=X_train, y=y_train, cv=10, n_jobs=1)
cross_val_score의 매개변수 estimator에 전달된 모델이 회귀일 때 KFold를 사용하고,
분류일 때는, StratifiedKFold를 사용한다.
cv 매개변수에 KFold등의 클래스 객체를 직접 전달할 수도 있다.
print('CV 정확도 점수: %s' %scores)
CV 정확도 점수: [0.93478261 0.93478261 0.95652174 0.95652174 0.93478261 0.95555556
0.97777778 0.93333333 0.95555556 0.95555556]
print('CV 정확도: %.3f +/- %.3f' %(np.mean(scores), np.std(scores)))
cross_val_score함수가 검증에 사용하는 측정 지표는 기본적으로 회귀일 때는 R^2,
분류일 때는 정확도이다.
scoring 매개변수를 지정하여 다른 지표로 변경할 수 있다.
cross_validate 함수를 사용하면 각 폴드에서 훈련과 테스트에 걸린 시간을 반환하고, scoreing 매개변수에 지정한 평가 지표마다
훈련 점수와 테스트 점수를 반환한다.(train_XXXX, test_XXXX 키를 이용해서 딕셔너리 값을 얻을 수 있다.
from sklearn.model_selection import cross_validate
scores=cross_validate(estimator=pipe_lr, X=X_train, y=y_train, scoring=['accuracy'], cv=10, n_jobs=-1)
print('CV 정확도 점수: %s' %scores['test_accuracy'])
CV 정확도 점수: [0.93478261 0.93478261 0.95652174 0.95652174 0.93478261 0.95555556
0.97777778 0.93333333 0.95555556 0.95555556]
print('CV 정확도: %.3f +/- %.3f' %(np.mean(scores['test_accuracy']), np.std(scores['test_accuracy'])))
cross_val_predict는 cross_val_score와 비슷한 인터페이스를 제공하지만,훈련 데이터셋의 각 샘플이 테스트 폴드가
되었을 때, 만들어진 예측을 반환한다.
따라서 corss_val_predict 함수를 사용해서 모델의 성능을 계산하면 cross_val_score와 결과가 다르며,
바람직한 일반화 성능 추정이 아니다.
cross_val_predict 함수는 훈련 데이터셋에 대한 예측 결과를 시각화 하거나 7장에서 소개하는 스태킹(Stacking)앙상블(ensemble)
방법처럼 다른 모델에 주입할 훈련 데이터를 만들기 위해 사용할 수 있다.
from sklearn.model_selection import cross_val_predict
preds=cross_val_predict(estimator=pipe_lr, X=X_train, y=y_train, cv=10, n_jobs=-1)
preds[:10]
array([0, 0, 0, 0, 0, 0, 0, 1, 1, 1])
method 매개변수에 반환될 값을 계산하기 위한 모델의 메서드를 지정할 수 있다.
method=‘predict_proba’로 지정하면, 예측 확률을 반환한다.
predict, predict_proba, predict_log_proba, decision_function 등이 사용 가능하며, default 값은 predict이다.
from sklearn.model_selction import cross_val_predict
preds=cross_val_predict(estimator=pipe_lr, X=X_train, y=y_train, cv=10, n_jobs=-1, method='predict_proba')
preds[:10]
>>> preds[:10]
array([[9.93982352e-01, 6.01764759e-03],
[7.64328337e-01, 2.35671663e-01],
[9.72683946e-01, 2.73160539e-02],
[8.41658121e-01, 1.58341879e-01],
[9.97144940e-01, 2.85506043e-03],
[9.99803660e-01, 1.96339882e-04],
[9.99324159e-01, 6.75840609e-04],
[2.12145074e-06, 9.99997879e-01],
[1.28668437e-01, 8.71331563e-01],
[7.76260670e-04, 9.99223739e-01]])
cross_val_scores기능은 각 폴드의 평가를 CPU 코어에 분산할 수 있다.
n_job를 통해서 사용할 CPU 코어 개수를 지정할 수 있으며, n_jobs=-1로 설정할 시 모든 CPU 코어를 사용하여 병렬 처리 한다.